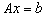
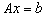
polega na zastąpieniu tego układu równoważnym, mnożąc układ wyjściowy przez odwrotność pewnej macierzy B, zwanej preconditionerem:
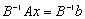 .
.Jeżeli teraz zaczniemy rozwiązywać powyższy układ metodą iteracyjną, np. metodą iteracji prostej to otrzymujemy następujące wzory na kolejne przybliżenia rozwiązania:
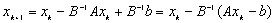 .
.Oznaczając 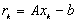 mamy:
mamy:
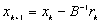 ,
,czyli w każdym kroku iteracyjnym mnożymy wektor residualny przez odwrotność macierzy B: po prostu rozwiązujemy układ z macierzą preconditionera. Od macierzy B żądamy:
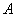 :
jak wiadomo, prędkość zbieżności metod iteracyjnych zazwyczaj silnie zależy
do współczynnika uwarunkowania macierzy układu; zmniejszenie tego współczynnika
poważnie wpływa na obniżenie liczby iteracji i tym samym - kosztu rozwiązania
całego układu równań; szczegóły można znaleźć w [6].
:
jak wiadomo, prędkość zbieżności metod iteracyjnych zazwyczaj silnie zależy
do współczynnika uwarunkowania macierzy układu; zmniejszenie tego współczynnika
poważnie wpływa na obniżenie liczby iteracji i tym samym - kosztu rozwiązania
całego układu równań; szczegóły można znaleźć w [6].SLES sles; PC pc; ... SLESCreate(MPI_COMM_WORLD,&sles); SLESGetPC(sles,&pc); ...
|
|
|
|
| Jacobi | PCJACOBI | jacobi |
| Blokowy Jacobi | PCBJACOBI | bjacobi |
| Blokowy GaussSeidel (tylko sekwencyjny) | PCBGS | bgs |
| SOR (i SSOR) | PCSOR | sor |
| SOR z trickiem Eisenstat'a | PCEISENSTAT | eisenstat |
| Niekompletny rozkład Choleskiego | PCICC | icc |
| Niekompletny rozkład LU | PCILU | ilu |
| Addytywna metoda Schwarza | PCASM | asm |
| Eliminacja Gaussa LU | PCLU | lu |
| Bez preconditionera | PCMINUS_ONE | minus_one |
| Zdefiniowany przez użytkownika | PCSHELL | shell |
SLES sles; KSP ksp; PC pc; ... SLESCreate(MPI_COM_WORLD,&sles); ... SLESGetKSP(sles,&ksp); KSPSetType(ksp,KSPPREONLY); SLESGetPC(sles,&pc); PCSetType(pc,LU); ... SLESSolve(sles);
... SLESCreate(MPI_COMM_WORLD,&sles); SLESGetPC(sles,&pc); PCSetType(pc,PCICC); ...
|
|
|
| PCASMGetSubSLES() | Wydobywa z preconditionera tablicę SLESów służących do rozwiązywania zadania w podobszarach. |
| PCASMSetLocalSubdomains() | Ustawia ile obszarów obsługuje procesor wywołujący tę funkcję. |
| PCASMSetOverlap() | Ustawia szerokość zakładki między obszarami. |
| PCASMSetTotalSubdomains() | Ustawia całkowitą liczbę podobszarów. |
| PCASMSetType() | Ustawia rodzaj metody Schwarza - sposób w jaki dane będą przekazywane między procesorami. |
#include "common.h"
static char help[] ="Preconditioner trywialny\n";
/*************************************************
Działanie preconditionera
*************************************************/
int Pierwszy_pc(void *ctx,Vec v0,Vec v1)
{
VecCopy(v0,v1);
return(0);
}
/*************************************************
Program glowny
*************************************************/
int main(int argc,char **args)
{
PetscInitialize(&argc,&args,PETSC_NULL,help);
Przygotuj_zadanie(N,&sles,&A,&u0,&u,&f);
/*ustawienie preconditionera powłokowego */
SLESGetPC(sles,&pc);
PCSetType(pc,PCSHELL);
PCShellSetApply(pc,Pierwszy_pc,PETSC_NULL);
PCShellSetName(pc,"Mój pierwszy preconditioner");
/*rozwiązanie zadania */
Rozwiaz(sles);
/*Zwalnianie pamięci*/
Zwolnij_pamiec(sles,A,u,u0,f);
PetscFinalize();
}
Po uruchomieniu mamy:
hydra:/usr/home/bin/> przyklad Iteracje = 351 Blad = 6.364062e-04 hydra:/usr/home/bin/>
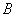 jest w tym wypadku macierzą identyczności.
jest w tym wypadku macierzą identyczności.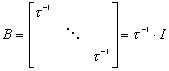 ,
,dla odpowiednio dobranej wartości parametru 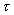 .
Jak wiadomo, najlepszy parametr metody Richardsona uzyskuje się, korzystając
z najmniejszej i największej wartości własnej macierzy (opis metody Richardsona
można znaleźć w [6]). Dlatego jako
kontekst podane zostaje oszacowanie na spektrum macierzy. Podobnie jak
w poprzednim przypadku, pliki common.h
i common.c są podane na końcu tego rozdziału.
.
Jak wiadomo, najlepszy parametr metody Richardsona uzyskuje się, korzystając
z najmniejszej i największej wartości własnej macierzy (opis metody Richardsona
można znaleźć w [6]). Dlatego jako
kontekst podane zostaje oszacowanie na spektrum macierzy. Podobnie jak
w poprzednim przypadku, pliki common.h
i common.c są podane na końcu tego rozdziału.
przyklad8_2.c
#include "common.h"
static char help[] ="Preconditioner Richardsona\n";
/***************************************************
Kontekst dla preconditionera
***************************************************/
typedef struct {
double lambdamin;
double lambdamax;} Kontekst;
Kontekst k_pc;
/***************************************************
Dzialanie preconditionera
***************************************************/
int Richardson_pc(void *ctx,Vec v0,Vec v1)
{
Scalar val=1;
Kontekst *kn=(Kontekst*)ctx;
val=2/(kn->lambdamin+kn->lambdamax);
VecCopy(v0,v1);
VecScale(&val,v1);
return(0);
}
/**************************************************
Program glowny
**************************************************/
main(int argc,char **args)
{
PetscInitialize(&argc,&args,PETSC_NULL,help);
Przygotuj_zadanie(N,&sles,&A,&u0,&u,&f);
SLESGetPC(sles,&pc);
PCSetType(pc,PCSHELL);
PCShellSetApply(pc,Richardson_pc,(void*)&k_pc);
PCShellSetName(pc,"Preconditioner Richardsona");
k_pc.lambdamin=1/N;
k_pc.lambdamax=1;
Rozwiaz(sles);
Zwolnij_pamiec(sles,A,u0,u,f);
PetscFinalize();
}
Po uruchomieniu mamy:
hydra:/usr/home/bin/> przyklad Iteracje = 80 Blad = 4.366761e-04 hydra:/usr/home/bin/>
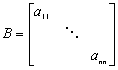
gdzie aii oznaczają elementy diagonalne macierzy zadania A. W implementacji tego preconditionera skorzystamy więc ze zmiennej kontekstowej: jako kontekst podamy główną diagonalę macierzy zadania. Zwróćmy uwagę na funkcję PrzygotujPreconditioner() - wykonuje on podobną pracę, co w standardowym przypadku funkcja PCSetUp().
przyklad8_3.c
#include "common.h"
static char help[] ="Preconditioner Jacobi'ego";
/***********************************************
Inicjalizacja kontekstu
preconditionera
***********************************************/
int PrzygotujPreconditioner(Mat A,Vec *k_pc)
{
int m,n;
MatGetSize(A,&m,&n);
If(m!=n)
{
PetscPrintf(MPI_COMM_WORLD,"Hej!!
Macierz nie jest kwadratowa\n");
}
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,m,k_pc);
MatGetDiagonal(A,(*k_pc));
return(0);
}
/***********************************************
Dzialanie preconditionera
***********************************************/
int wlasny_pc(void *ctx,Vec v0,Vec v1)
{
Vec *k_pc=(Vec*)ctx;
/* Dzialanie preconditionerem Jacobiego */
VecPointwiseDivide(v0,*k_pc,v1);
return(0);
}
/***********************************************
Program glowny
***********************************************/
int main(int argc,char **args)
{
Vec diag;
PetscInitialize(&argc,&args,PETSC_NULL,help);
Przygotuj_zadanie(N,&sles,&A,&u0,&u,&f);
SLESGetPC(sles,&pc);
PCSetType(pc,PCSHELL);
PrzygotujPreconditioner(A,&diag);
PCShellSetApply(pc,wlasny_pc,&diag);
PCShellSetName(pc,"Preconditioner Jacobi'ego");
Rozwiaz(sles);
PetscFinalize();
}
Po uruchomieniu mamy:
hydra:/usr/home/bin/> przyklad Iteracje = 63 Blad = 4.364761e-04 hydra:/usr/home/bin/>
#include "common.h"
static char help[] ="Addytywny preconditioner
Schwarza z grubą siatką
Marcin Bojko";
/* Tutaj definiujemy strukture, która bedzie
kontekstem preconditionera */
typedef struct {
Mat I,Ag;
/* macierz interpolacji i zadania na grubej siatce */
int rozmiar,mg;
/* mg to rozmiar grubej siatki*/
int m,M,zakl;
/* M to ilość podobszarów w jednym kierunku */
/* wszystkich podobszarów będzie M*M */
IS *boxy;
/* tu będzie składowana dekompozycja */
Vec fg,ug;
/* pomocnicze wektory zadania na grubej siatce */
Scalar value[3],val;
SLES slesg;
/* SLES dla zadania na gubej siatce */
PC pc_box;
/* preconditioner Schwarza na boxach */
} asm_ctx;
int Preconditioner(void *pointer,Vec u0,Vec
u1);
void Przygotuj_macierz_interpolacji(asm_ctx
*ctx)
{
int i,j,row,col,r,k,l;
int mg=ctx->mg,m=ctx->m,rozmiar=ctx->rozmiar;
Scalar val;
MatCreate(MPI_COMM_WORLD,rozmiar*rozmiar,mg*mg,&ctx->I);
for(j=0;j<mg;j++)
for(i=0;i<mg;i++)
{
col=i+mg*j;
row=r=(m-1)*rozmiar+m-1+i*m+j*m*rozmiar;
val=1;
MatSetValues(ctx->I,1,&row,1,&col,&val,INSERT_VALUES);
for(k=1;k<m;k++)
{
val=(Scalar)(m-k)/m;
for(l=0;l<6*k;l++)
{
if((l>1)&&(l<k))row=r-k*rozmiar+l;
if((l>k-1)&&(l<2*k))row=r-k*rozmiar+k+(l-k)*rozmiar;
if((l>2*k-1)&&(l<3*k))row=r+k+(l-2*k)*(rozmiar-11);
if((l>3*k-1)&&(l<4*k))row=r+k*rozmiar-(l-3*k);
if((l>4*k-1)&&(l<5*k))row=r+k*(rozmiar-1)-(l-4*k)*rozmiar;
if((l>5*k-1)&&(l<6*k))row=r-k-(l-5*k)*(rozmiar-1);
MatSetValues(ctx->I,1,&row,1,&col,&val,INSERT_VALUES);
}
}
}
MatAssemblyBegin(ctx->I,MAT_FINAL_ASSEMBLY);
MatAssemblyEnd(ctx->I,MAT_FINAL_ASSEMBLY);
MatCompress(ctx->I);
}
Przygotuj_preconditioner(asm_ctx *ctx,PC *pc_shell)
{
int flag;
int rozmiar=ctx->rozmiar,M=ctx->M;
int zakl=ctx->zakl,mg=ctx->mg;
PC pcg;
KSP ksp;
/*Przygotowanie preconditionera Schwarza
*/
PCCreate(MPI_COMM_WORLD,&ctx->pc_box);
PCSetType(ctx->pc_box,PCASM);
PCASMCreateSubdomains2D(rozmiar,rozmiar,M,M,1,zakl,&i,&ctx->boxy);
PCASMSetLocalSubdomains(ctx->pc_box,i,ctx->boxy);
PCSetOperators(ctx->pc_box,A,A,SAME_PRECONDITIONER);
PCSetVector(ctx->pc_box,u);
PCASMSetOverlap(ctx->pc_box,0);
PCSetUp(ctx->pc_box);
/* gruba siatka */
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,mg*mg,&ctx->fg);
VecDuplicate(ctx->fg,&ctx->ug);
Przygotuj_macierz_laplasjanu(&ctx->Ag,mg,mg);
SLESCreate(MPI_COMM_WORLD,&ctx->slesg);
SLESSetOperators(ctx->slesg,ctx->Ag,ctx->Ag,SAME_PRECONDITIONER);
SLESGetPC(ctx->slesg,&pcg);
PCSetType(pcg,PCLU);
SLESGetKSP(ctx->slesg,&ksp);
KSPSetType(ksp,KSPPREONLY);
Przygotuj_macierz_interpolacji(ctx);
}
int main(int argc,char **args)
{
KSP ksp;
PC pc_shell;
asm_ctx ams;
double norma,emin,emax;
int i,flag;
PetscInitialize(&argc,&args,PETSC_NULL,help);
ams.M=4;
ams.mg=7;
ams.zakl=1;
ams.m=8;
OptionsGetInt(PETSC_NULL,"M",&ams.M,&flag);
OptionsGetInt(PETSC_NULL,"mg",&ams.mg,&flag);
OptionsGetInt(PETSC_NULL,"m",&ams.m,&flag);
OptionsGetInt(PETSC_NULL,"zakl",&ams.zakl,&flag);
ams.rozmiar=(ams.mg+1)*ams.m-1;
Przygotuj_zadanie(ams.rozmiar,&sles,&A,&u0,&u,&f);
SLESGetKSP(sles,&ksp);
SLESGetPC(sles,&pc_shell);
Przygotuj_preconditioner(&ams,&pc_shell);
PCSetType(pc_shell,PCSHELL);
PCShellSetApply(pc_shell,Preconditioner,&ams);
PCShellSetName(pc_shell,"asmbox");
KSPSetType(ksp,KSPGMRES);
KSPSetComputeSingularValues(ksp);
SLESSetFromOptions(sles);
Rozwiaz(sles);
KSPComputeExtremeSingularValues(ksp,&emax,&emin);
PetscFPrintf(MPI_COMM_WORLD,stderr,"
Uwarunkowanie = %e\n\n",emax/emin);
SLESDestroy(ams.slesg);
MatDestroy(ams.Ag);
VecDestroy(ams.ug);
VecDestroy(ams.fg);
MatDestroy(ams.I);
Zwolnij_pamiec(sles,A,u0,u,f);
PetscFinalize();
}
int Preconditioner(void *pointer,Vec v0 ,Vec
v1)
{
asm_ctx *ams=(asm_ctx*)pointer;
int its;
/*rozwiązanie na boxach */
PCApply(ams->pc_box,v0,v1);
/* rozwiazanie na grubej siatce */
MatMultTrans(ams->I,v0,ams->fg);
SLESSolve(ams->slesg,ams->fg,ams->ug,&its);
VecScale(&val,ams->ug);
MatMultAdd(ams->I,ams->ug,v1,v1);
return(0);
}
common.h #include "sles.h" Vec u,u0,f; Mat A; SLES sles; PC pc; int N=100,its,i; Scalar val; double norma; int Rozwiaz(SLES sles); int Przygotuj_zadanie(int rozmiar,SLES *sles,Mat *A,Vec *u0,Vec *u,Vec *f); int Zwolnij_pamiec(SLES sles,Mat A,Vec u0,Vec u,Vec f); int Przygotuj_macierz_laplasjanu(Mat *A,int roz_x,int roz_y);makefile (do skompilowania powyższych przykładów)
common.c #include "common.h" int Przygotuj_macierz_laplasjanu(Mat *A,int roz_x,int roz_y) { Scalar val; int n,m,i,j; n=roz_x*roz_y; MatCreateSeqAIJ(MPI_COMM_WORLD,n,n,5,PETSC_NULL,A);; for(i=0;i<n;i++) { val=4; MatSetValues(*A,1,&i,1,&i,&val,INSERT_VALUES);; /* elementy na dalekich kodiagonalach */ val=-1; j=i+roz_y; if(j<n) { MatSetValues(*A,1,&i,1,&j,&val,INSERT_VALUES);; MatSetValues(*A,1,&j,1,&i,&val,INSERT_VALUES);; } /* elementy na bliskich kodiagonalach */ j=i+1; val=-1; if(j<n)if(((i+1)%roz_x)!=0||(i==0)) { MatSetValues(*A,1,&i,1,&j,&val,INSERT_VALUES);; MatSetValues(*A,1,&j,1,&i,&val,INSERT_VALUES);; } } MatAssemblyBegin(*A,MAT_FINAL_ASSEMBLY); ; MatAssemblyEnd(*A,MAT_FINAL_ASSEMBLY); ; MatCompress(*A); return(0); } /*Przygotowanie zadania testowego */ int Przygotuj_zadanie(int N,SLES *sles,Mat *A,Vec *u0,Vec *u,Vec *f) { PetscRandom rctx; VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,N*N,f); VecDuplicate(*f,u); VecDuplicate(*f,u0); Przygotuj_macierz_laplasjanu(A,N,N); SLESCreate(MPI_COMM_WORLD,sles); SLESSetOperators(*sles,*A,*A,SAME_PRECONDITIONER); PetscRandomCreate(MPI_COMM_WORLD,RANDOM_DEFAULT,&rctx); VecSetRandom(rctx,*u0); PetscRandomDestroy(rctx); MatMult(*A,*u0,*f); return(0); } /* Zwolnienie pamięci po zadaniu testowym */ int Zwolnij_pamiec(SLES sles,Mat A,Vec u0,Vec u,Vec f) { SLESDestroy(sles); VecDestroy(u0); VecDestroy(u); VecDestroy(f); MatDestroy(A); } int Rozwiaz(SLES sles) { SLESSetFromOptions(sles); SLESSolve(sles,f,u,&its); /*Podanie wynikow*/ val=-1; VecAXPY(&val,u,u0); VecNorm(u0,NORM_2,&norma); PetscFPrintf(MPI_COMM_WORLD,stderr,"\nLiczba iteracji = %i\n",its); PetscFPrintf(MPI_COMM_WORLD,stderr," Blad = %e\n",(double)norma); }
include $(PETSC_DIR)/bmake/$(PETSC_ARCH)/base CC = gcc -DPETSC_LOG CLINKER = gcc CFLAGS = $(PCONF) $(PETSC_INCLUDE) $(BS_INCLUDE) LIBBASE = libpetscsles libpetscsnes OPTS = -O BOPT = O wszystko: przykład8_1 przykład8_2 przykład8_3 przykład8_4 przykład8_1: przyklad8_1.o $(CLINKER) -o przyklad8_1 przyklad8_1.o common.o $(PETSC_LIB) $(RM) przyklad8_1.o przyklad8_2: przyklad8_2.o $(CLINKER) -o przyklad8_2 przyklad8_2.o common.o $(PETSC_LIB) $(RM) przyklad8_2.o przyklad8_3: przyklad8_3.o $(CLINKER) -o przyklad8_3 przyklad8_3.o common.o $(PETSC_LIB) $(RM) przyklad8_3.o przyklad8_4: przyklad8_4.o $(CLINKER) -o przyklad8_4 przyklad8_4.o common.o $(PETSC_LIB) $(RM) przyklad8_4.o
hydra:/usr/home/students/bojko/>przyklad8_1
Można też od razu skompilować wszystkie programy (pod warunkiem, że wszystkie pliku źródłowe są w jednym katalogu) poleceniem:
hydra:/usr/home/students/bojko/>make wszystko
 |
 |
 |